真核生物中,Cdc45-MCM-GINS (CMG解旋酶)与DNA聚合酶(Polε)的紧密耦合,是驱动复制起始、维持分叉进程的前提。然而,二者在复制叉上的活动机制在很大程度上尚未可知。
近日,香港大学生物科学学院领衔在Nature Communication上发表了题为Synergism between CMG helicase and leading strand DNA polymerase at replication fork的研究文章。通过对酵母前导链复制体的一系列研究,前所未有地阐明了一种CMG解旋酶与Polε的协同工作机制,及其协调DNA解螺旋及合成的动态图景。

1 酵母前导链复制体的整体结构
作者组装了一个前导链复制体复合物,并用低温电镜技术进行研究。结果显示,在此复制体结构中,CMG解旋酶作为核心,将Tof1-Csm3组装到MCM环Mcm6/4/7侧的NTD面,将Ctf4同源三聚体组装到Cdc45和GINS的界面上,而Polε则与MCM环Mcm2/5/3侧的GINS和CTD面结合(补充图2a-e)。从MCM环的NTD层伸出的亲本双链DNA被Tof1-Csm3捕获,并向Mcm6/4/7侧弯曲(补充图2a, c, d, f)。
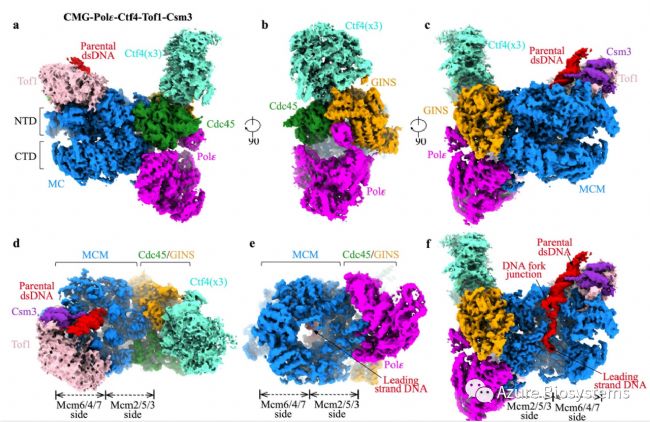
补充图2. 前导链复制体的总体结构(a-c)DNA复制叉上CMG-Polε-Ctf4-Tof1-Csm3复制体的冷冻电镜密度图(侧视图)。(d,e)复制体MCM环的顶部NTD(d)和底部CTD(e)视图。(f) 与(c)相同,但去除Mcm3和Mcm7以突出MCM环内叉DNA的构象。
2 Polε与CMG MCM环的docking
在复制体结构中,Polε通过四个对接位点与CMG相互作用(图1a-d)。在对接位点(DS) 1上,Polε全酶高柔韧性的Dpb2-NTD(残基12-98)稳定地锚定在GINS Psf1(残基161-208)的b结构域上。
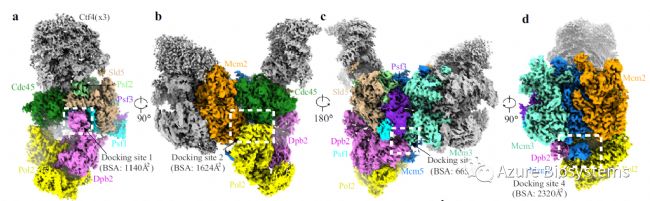
图1. Polε与CMG解旋酶互作详情图。a–d复制体的冷冻电镜密度图(侧视图)。
然而, Polε可能不依赖于Dpb2-NTD而被整合到复制体中。作者构建了一个Dpb2-NTD 敲除的Polε突变体(称为PolεΔ2N)。体外拉下实验(Sapphire激光扫描成像系统检测)显示,尽管亲和力低于野生型,PolεΔ2N仍可与DNA-CMG复合体进行作用(Polε-WT, 图2a,泳道 6-7)。可见MCM环介导了Polε与CMG解旋酶的直接结合,而不依赖于Dpb2-NTD。
有趣的是,在没有DNA的情况下,PolεΔ2N与CMG复合体的结合受到严重抑制(图2a,泳道11);然而Polε-WT则表现出很强的CMG结合亲和力(图2a,泳道10)。此时,Dpb2-NTD与Psf1的相互作用,成为Polε与CMG耦合的必要条件。同时也表明,分叉DNA在调节Polε进入前导链复制体中具有关键作用。
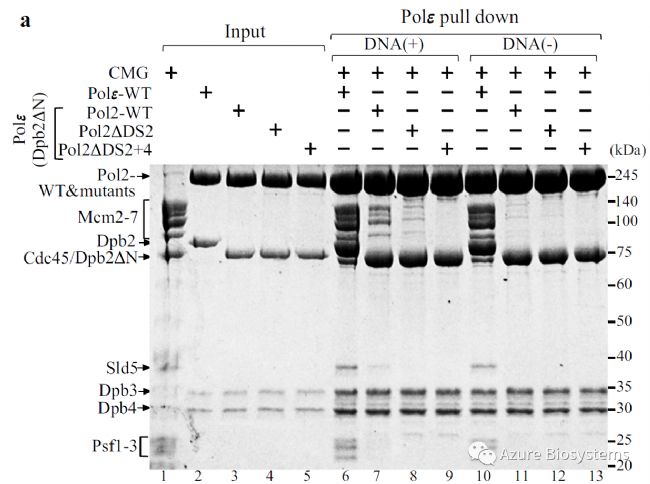
图2a. CMG解旋酶体外拉下实验(Sapphire激光扫描成像系统拍摄)
3 polε- MCM耦合是复制起始的必要条件
作者构建了一系列删除MCM结合域的pol2突变体。结果显示,在限制性条件下,同时敲除DS2和DS4(pol2ΔDS2+4)将导致pol2-iAID细胞(可条件性耗尽内源Pol2- aid融合蛋白)无法存活。
作者接着进行体外拉下实验(Sapphire激光扫描成像系统,图2a,泳道4-5)。结果显示, Pol2ΔDS2突变体中, PolεΔ2N与DNA-CMG复合体的亲和力显著降低(图2a, 泳道 8), Pol2ΔDS2+4中几乎完全消失(图2a,泳道9)。
在耗尽内源性pol2-aid蛋白后,进行Psf2-Flag免疫沉淀分析。在POL2-WT细胞中,与Psf2-Flag共沉淀的Mcm2和Cdc45仅出现在S期早期;在pol2ΔDS2突变体(图2e, 泳道5-6)中,共沉淀水平降低;在pol2ΔDS2+4突变体(图2e, 泳道7-8)中则被抑制。
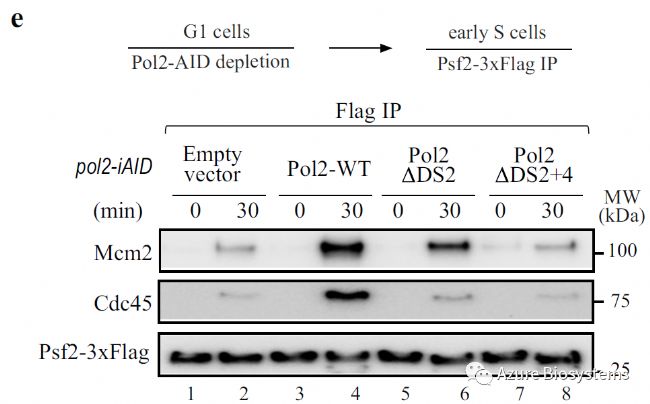
图2e. Psf2-Flag免疫沉淀实验 (Sapphire激光扫描成像系统拍摄)
可见,DS2和DS4对于稳定Polε与MCM环的结合至关重要,稳定的polε- MCM耦合是CMG形成驱动复制起始的必要条件。
此外,体外解旋酶实验中(Sapphire激光扫描成像系统检测),无Polε存在时,CMG解旋酶可有效解螺旋分叉的双链DNA (图6b, d),Polε存在时,只表现出轻微的CMG活性抑制(图6c, d),表明Polε结合对CMG活性几乎没有影响。
体外扩增实验(Sapphire激光扫描成像系统检测)显示,与polε-WT的高水平全长产物相比,PolεΔDS2的复制效率降低,PolεΔDS2+4没有明显的DNA合成(图6g)。不加CMG的对照组中,只有极低水平的DNA合成产物。说明Polε与CMG解旋酶的耦合有助于在前导链的复制延申。
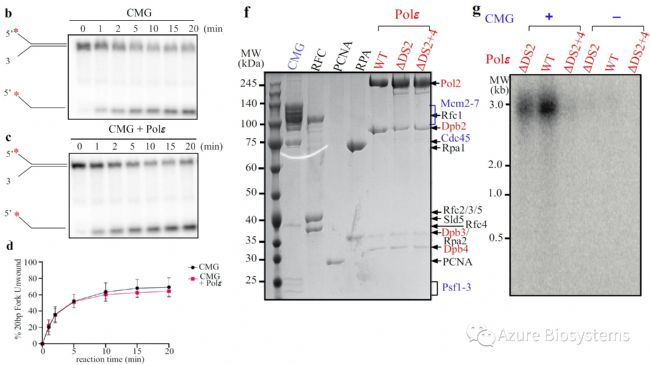
图6. b-c. 有无Polε情况下,CMG解旋酶的非变形PAGE分析 d. 复制叉解螺旋效果 f. 纯化的DNA复制相关因子,SDS-PAGE考染显色分析 g. 复制产物的琼脂糖凝胶实验(Sapphire激光扫描成像系统拍摄)
综上所述,本研究阐明了复制体的一种内在机制,通过协调CMG和Polε的活动,能够解决复制叉易位过程中的各种障碍、DNA损伤及表观遗传标记。这种解旋酶与DNA聚合酶的周期性耦合机制,或是生物体用于高保真复制基因组的有效策略。
原文链接:https://doi.org/10.1038/s41467-023-41506-0
Azure Biosystems
Azure Biosystems成立于2013年,总部位于美国加州湾区,是一家致力于生命科学领域先进生物成像系统研发、制造、推广及服务一体的创新型公司。拥有分子成像领域近30年经验的卓越团队,Azure公司革新推出的Azure系列新一代多功能分子成像系统和Sapphire激光扫描成像系统已成为技术领先的特色产品。目前,公司已形成分子凝胶成像、Western Blot、RT-PCR及配套试剂耗材等产品线。
公司成像设备全球装机>5000台,用户数量>30000,包括美国最高科研机构NIH、梅约诊所、哈佛大学、中国科学院、清华大学、诺华制药等知名科研院所、医院及生物医药企业等。截至目前,发表文章>3000篇,发表刊物包括Cell、Science、Nature三大期刊及子刊、JBC、JMC等国际著名期刊杂志。


